유전체 공부를 하다보면 SNP chip data를 사용하는게 아닌, NGS 또는 GWAS를 통해 알 수 있는 정보들로 Pathogenicity 판단을 할 때 쓸 수 있는 가이드라인이 있다.
병원에서 유전자 검사라고 통칭되는 검사를 하면 받아보는 항목이 이걸 기반으로 한다.
정말 대단한 집단지성의 힘이 아닐 수 없다...
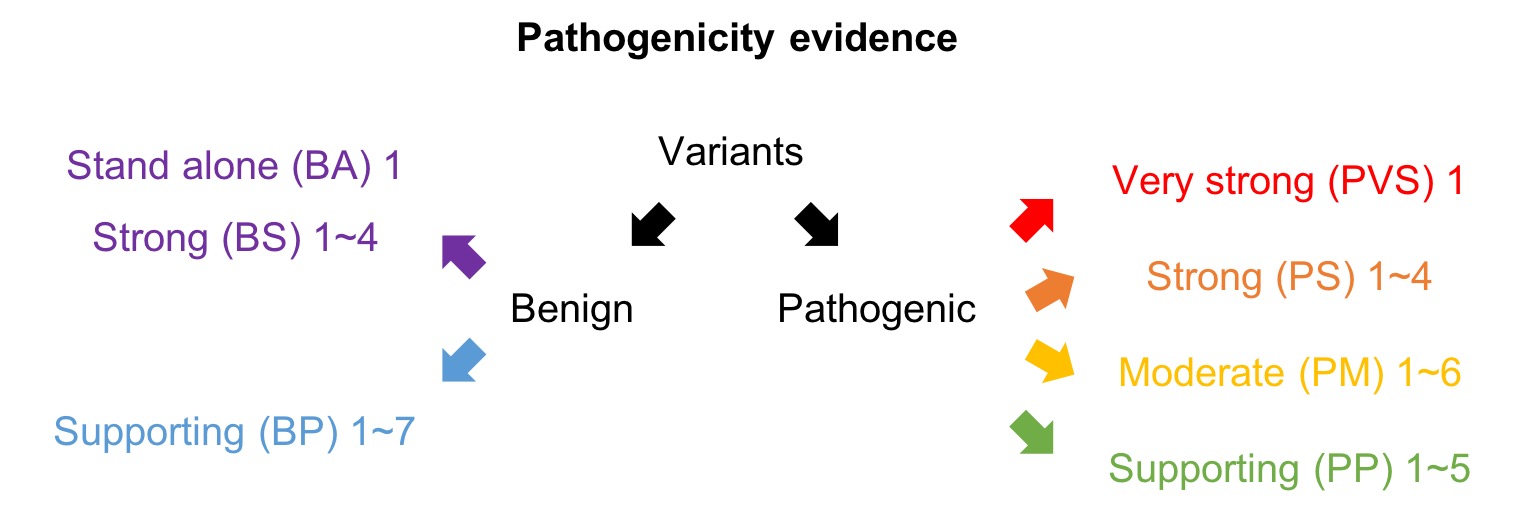

ACMG (American College of Medical Genetics and Genomics) 가이드라인에서 pathogenicity (병원성) 판단을 할 때 사용하는 데이터는 DNA chip (SNP array) 데이터가 아니라 주로 NGS (Next-Generation Sequencing) 기반의 전장유전체 또는 타겟 유전자 데이터.
1. ACMG 가이드라인에서 병원성 평가에 사용하는 데이터 종류
ACMG 가이드라인(특히 2015 ACMG-AMP 기준)에서는 특정 변이가 **pathogenic (병원성)**인지, **likely pathogenic (가능성 높은 병원성)**인지 등을 판단할 때 여러 가지 증거를 사용합니다. 이때 고려하는 데이터는 다음과 같습니다.
✅ 유전체 분석 데이터 (NGS, Sanger sequencing)
- Whole-Exome Sequencing (WES, 전장 엑솜 시퀀싱)
- Whole-Genome Sequencing (WGS, 전장 유전체 시퀀싱)
- Targeted Gene Panel (특정 유전자 패널 시퀀싱)
- Sanger sequencing (특정 유전자 검증용)
→ 이러한 기법들은 유전자의 염기서열을 직접 분석하기 때문에 특정 변이가 단일염기 변이(SNV), 인델(InDel), copy number variation (CNV) 등으로 인해 단백질 기능에 영향을 미치는지를 확인할 수 있음.
✅ 기능 연구 데이터 (Functional Studies)
- 세포주, 동물 모델 실험 등에서 변이가 특정 단백질 기능에 미치는 영향을 확인
- 예: 특정 변이가 단백질 활성도를 낮추거나 세포의 생존율에 영향을 미치는지 분석
✅ 인구집단 데이터 (Population Data)
- gnomAD, ExAC, 1000 Genomes, KRGDB (Korean Reference Genome Database) 등 대규모 데이터베이스에서 변이 빈도를 분석
- **빈도가 낮은 변이(rare variant)**가 질병과 연관될 가능성이 높음
- 예: 병원성 변이는 보통 일반 인구에서 매우 희귀함
✅ 유전병 데이터베이스 (ClinVar, HGMD, OMIM 등)
- 특정 변이가 이전에 보고된 질병과의 연관성이 있는지 확인
- ClinVar에서 pathogenic 또는 likely pathogenic으로 분류된 변이는 임상적으로 중요할 가능성이 큼
✅ 단백질 구조 및 예측 알고리즘 (In Silico Prediction)
- SIFT, PolyPhen-2, MutationTaster 같은 소프트웨어를 이용해 변이가 단백질 기능을 어떻게 변화시키는지 예측
- 예: 특정 변이가 아미노산 치환을 유발하여 단백질 구조를 불안정하게 만들 가능성이 있는지 분석
2. DNA Chip (SNP array) 데이터와 Pathogenicity 분석의 차이점
❌ DNA Chip 데이터는 ACMG 병원성 판단에 직접 활용되지 않음
DNA Chip 데이터는 SNP (단일 염기 다형성) 기반으로 미리 알려진 유전 변이를 탐지하는 방식입니다. 즉, **새로운 변이(rare variant)**를 발견하는 데 최적화되지 않았습니다.
🚨 DNA Chip이 병원성 변이를 찾기에 적절하지 않은 이유:
- SNP array는 미리 정의된 변이만 탐지 → 드문 병원성 변이를 놓칠 가능성이 큼
- 구조 변이(CNV), 희귀 변이(rare variant) 탐지가 어려움
- de novo 변이(새로운 변이) 확인 불가
- 단백질 기능 예측을 위한 full-sequence 정보 부족
✅ NGS가 더 적절한 이유
- 새로운 변이 검출 가능 (de novo 변이 포함)
- 단백질 코딩 영역 전체 분석 가능 (WES, WGS)
- SNP뿐만 아니라 인델, CNV, splicing 변이 등 탐지 가능
- Full sequence 정보 기반으로 in silico 예측 가능
3. 그럼 DNA Chip 데이터는 어디에 유용할까?
ACMG 가이드라인에서 말하는 pathogenicity 분석은 주로 NGS 데이터를 기반으로 하지만, DNA Chip 데이터도 일부 연구에 활용될 수 있음.
✅ DNA Chip (SNP array) 데이터 활용 가능 분야
- GWAS (Genome-Wide Association Study)
- 대규모 코호트 연구에서 특정 SNP와 질병의 연관성을 분석
- 병원성 평가가 아닌, 질병 위험도(risk score) 예측 목적
- 예: 특정 SNP가 뇌졸중 위험도를 높이는지 분석
- Polygenic Risk Score (PRS)
- 여러 SNP의 조합을 이용해 질병 위험도를 예측하는 방법
- 특정 유전자의 병원성 여부를 판단하는 것과는 다름
- 유전적 혈통 분석, 집단 유전학 연구
- 인구집단 내 유전자 빈도 확인
📌 즉, DNA Chip 데이터는 병원성 판단(pathogenicity classification)보다는 GWAS나 PRS 같은 연구에 더 적합함.
Pathogenicity 분석을 하려면 WES, WGS 같은 NGS 데이터가 필요함.
4. 결론
- ACMG 가이드라인에서 병원성 판단은 DNA Chip 데이터가 아니라 NGS 기반 데이터를 사용
- DNA Chip (SNP array) 데이터는 병원성 변이 탐지에 한계가 많음 (구조 변이, 희귀 변이 탐지 어려움)
- Pathogenicity를 정확히 평가하려면 WES/WGS, 기능 연구, 인구집단 데이터, 유전병 데이터베이스 등이 필요
- DNA Chip 데이터는 GWAS, PRS 등 대규모 유전 연구에 유용하지만, 개별 변이의 병원성 판별에는 적합하지 않음
결국, ACMG 가이드라인을 적용하여 병원성 변이를 판별하려면 NGS 데이터가 필수적이며, DNA Chip 데이터만으로는 이를 판단할 수 없습니다.
🔍 NGS가 DNA Chip보다 더 우수한 이유
비교 항목NGS (WES/WGS)DNA Chip (SNP Array)| 분석 범위 | 전장 유전체(WGS), 전장 엑솜(WES), 특정 유전자 패널 분석 | 미리 정해진 특정 SNP 위치만 분석 |
| 변이 탐지 능력 | SNV, Indel, CNV, 구조 변이, Splicing 변이 가능 | 주로 SNP 탐지 (Indel, CNV 분석 한계) |
| 새로운 변이 발견 | 가능 (de novo 변이 포함) | 불가능 (사전 정의된 SNP만 탐지) |
| 단백질 기능 예측 | 가능 (코딩 영역, 스플라이싱 예측 포함) | 불가능 (단일 염기 변이만 확인) |
| 질병과의 직접적인 연관 분석 | 가능 (병원성 변이 평가) | 주로 질병 연관 SNP 분석 (GWAS, PRS) |
| 비용 | 상대적으로 고가 (특히 WGS) | 상대적으로 저렴 |
✅ NGS가 분석할 수 있는 주요 변이 유형
NGS는 단순한 SNP(단일염기변이) 뿐만 아니라 더 다양한 변이를 탐지할 수 있어.
- SNP (Single Nucleotide Polymorphism)
- 단일 염기 치환 (ex: A → G 변이)
- DNA chip도 탐지 가능하지만, NGS는 더 많은 범위의 변이 탐지 가능
- Indel (Insertion/Deletion)
- 작은 삽입/결실 변이 (1~50bp)
- 코딩 영역에 존재하면 프레임시프트(Frameshift)로 큰 단백질 변화 유발 가능
- DNA chip으로는 탐지 불가능하지만, NGS는 쉽게 탐지 가능
- CNV (Copy Number Variation)
- 특정 유전자 또는 염색체 일부가 증가(duplication) 또는 감소(deletion)
- 특정 질환(예: 신경발달장애, 암)과 관련 있음
- NGS(WGS)에서는 정확한 CNV 분석 가능, 하지만 SNP array에서는 해상도가 낮아 정밀 분석이 어려움
- Splicing 변이
- 인트론과 엑손 경계에서 발생하여 RNA 스플라이싱에 영향을 미치는 변이
- 단순 SNP 기반 DNA chip으로는 감지 불가능
- NGS(WES/WGS)는 스플라이싱 예측 알고리즘을 통해 가능성 평가 가능
- Structural Variants (SV, 구조 변이)
- 큰 염색체 결실/중복, 역위(Inversion), 전좌(Translocation) 등
- DNA chip에서는 탐지 불가능하지만 WGS에서는 탐지 가능
- Mitochondrial DNA 변이
- NGS(WGS)에서만 가능 (DNA chip으로는 분석 불가)
💡 그래서 언제 NGS vs. DNA Chip을 사용할까?
✅ NGS를 사용해야 하는 경우
- 희귀 유전병 진단
- 새로운 병원성 변이 찾기 (WES/WGS 필요)
- 종양 유전체 분석 (암 돌연변이 탐색)
- Driver mutation, Tumor mutational burden(TMB) 분석
- 가계 내 변이 분석 (De novo 변이 분석)
- 부모에게 없는 새로운 변이가 환자에게 발생했는지 확인 가능
- CNV, 구조 변이(SV) 분석
- WGS 필요
✅ DNA Chip을 사용해야 하는 경우
- 대규모 GWAS 연구 (유전적 연관성 연구)
- 특정 질병과 관련된 SNP 찾기
- Polygenic Risk Score(PRS) 연구
- 유전적 계통 분석 (Population genetics)
- 인구집단 내 특정 SNP 빈도 확인
- 비용이 제한적인 경우
- NGS보다 저렴함
📌 결론: NGS는 훨씬 더 강력한 분석 도구
DNA Chip(SNP Array)은 미리 정의된 SNP를 확인하는 용도로는 유용하지만, 병원성 변이 분석에는 한계가 많음.
반면, NGS(WES/WGS)는 더 광범위한 유전체 변이를 분석할 수 있어 pathogenicity 판단에 적합하지!
✔ 병원성 변이(rare variant) 분석 → NGS(WES/WGS) 필수
✔ 대규모 GWAS 연구, PRS 계산 → DNA Chip(SNP Array) 유용
🚀 결국, 연구 목적에 따라 NGS와 DNA Chip 중 적절한 방법을 선택하는 것이 중요해!
'GENETICS' 카테고리의 다른 글
| 유전학에서 Additive Feature와 Dominance Feature 이해하기 (0) | 2025.02.28 |
|---|---|
| 헷갈리는 유전체 숫자? 한 번에 정리해 드립니다! (0) | 2025.02.24 |
| Exon to Genome (2) (0) | 2024.11.06 |
| Exon to Genome (1) (6) | 2024.11.05 |
| ped file (0) | 2024.09.30 |



댓글